Los avances recientes en el campo de la biología molecular de los sarcomas pediátricos, en especial el descubrimiento y caracterización de translocaciones cromosómicas específicas, han sentado las bases para la implantación de nuevas herramientas diagnósticas. En esta revisión se repasan las principales translocaciones asociadas a tumores pediátricos y se resumen sus características moleculares en relación a su capacidad oncogénica, su posible utilidad como herramientas de diagnóstico diferencial así como su posible relación con parámetros clínicos.
Recent advances in the knowledge of the molecular biology of paediatric sarcomas, especially the characterisation of chromosomal translocations associated specifically with particular types of cancer, have established bases for the introduction of new diagnostic tools. This article reviews the main chromosomal translocations associated with paediatric tumours, and summarises their molecular characteristics regarding their oncogenic capabilities, possible usefulness as a differential diagnostic tools and possible correlation with clinical parameters.
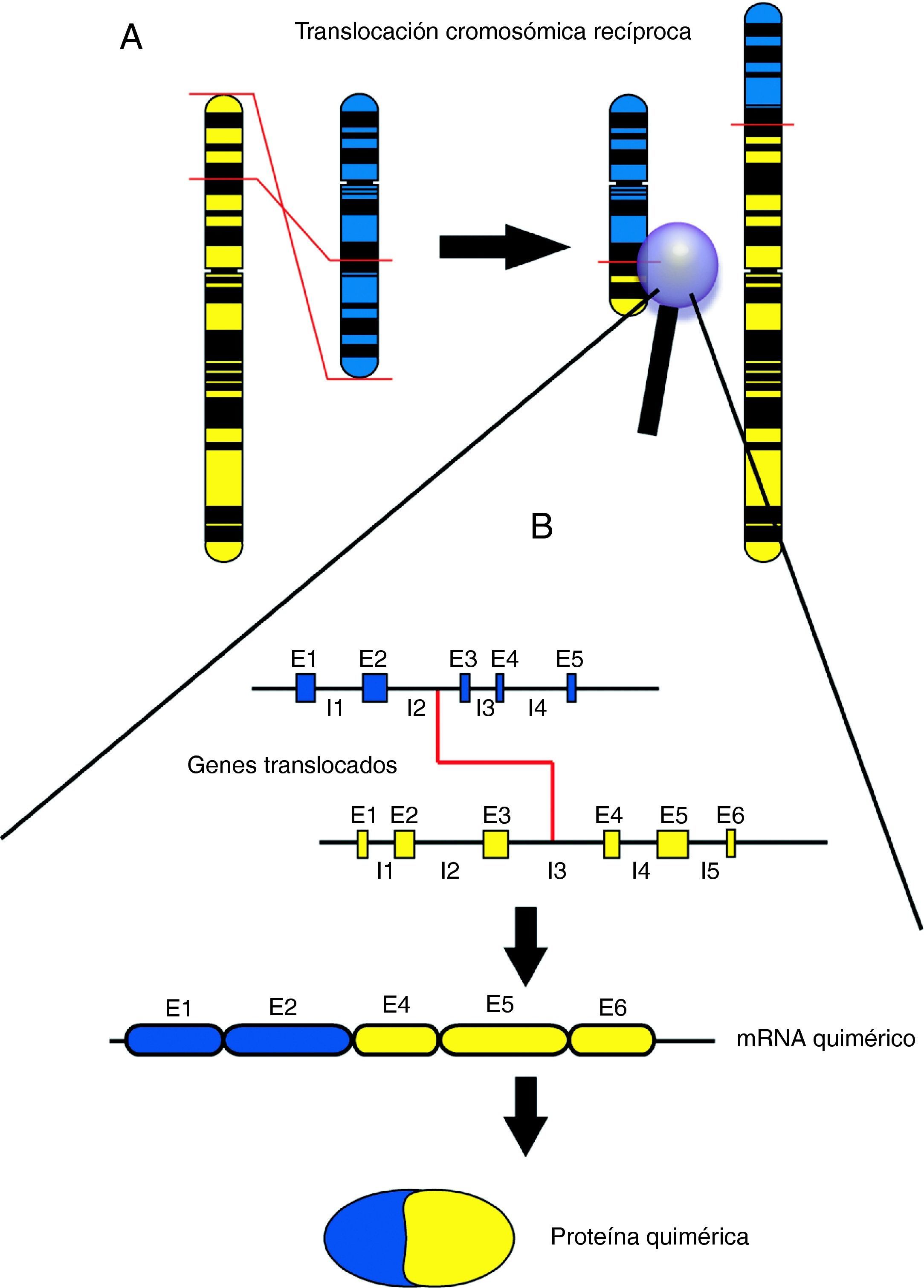
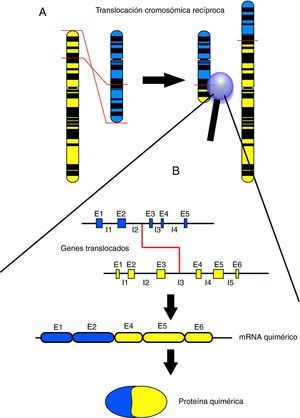
Durante las dos últimas décadas, el avance en el conocimiento de la biología molecular de los tumores pediátricos ha permitido descubrir la presencia de translocaciones cromosómicas asociadas específicamente a determinadas neoplasias. Las translocaciones cromosómicas implican el intercambio de fragmentos de ADN entre dos cromosomas y pueden dar lugar a fusiones entre dos genes, produciéndose en este caso genes de fusión que podrán dar lugar a su vez a proteínas aberrantes, generalmente denominadas quimeras o proteínas quiméricas (el término quimera tiene su origen en la palabra griega Khimaira, que en la mitología griega designaba un monstruo compuesto de partes de varios animales: parte superior de león, cola acabada en cabeza de serpiente y una cabeza de cabra surgida del centro de la espalda). Las translocaciones recíprocas dan lugar a dos productos de fusión, uno en cada cromosoma implicado (fig. 1). A pesar de la formación de estos dos productos, generalmente se detecta únicamente la quimera procedente de una de las dos fusiones, mientras que el producto recíproco suele ser indetectable o extremadamente escaso1–3, ya sea porque no llega a ser transcrito y/o traducido o bien a que el producto proteico no tiene estabilidad suficiente y es rápidamente degradado. En las translocaciones asociadas a cáncer, la proteína quimérica resultante suele conservar la funcionalidad original de al menos una de las proteínas implicadas en la fusión, pero su grado de activación suele ser excesivo y/o anómalo. Por este motivo, las proteínas quiméricas asociadas a cáncer presentan en la mayoría de casos una elevada actividad transformadora de las células que la sufren, promoviendo una intensa activación de los mecanismos de la oncogénesis. Al menos uno de los genes implicados en las translocaciones oncogénicas suele tener capacidad activadora de vías de transducción de señal o bien tiene la capacidad de activar la transcripción de genes pro-oncogénicos4,5, bien sea activando la proliferación celular, inhibiendo la diferenciación, evadiendo la apoptosis o activando mecanismos de movilidad e invasión.
A: Visión esquemática de una translocación recíproca a nivel cromosómico. Un fragmento del cromosoma amarillo se intercambia por un fragmento del cromosoma azul. B: Visión esquemática del punto de corte de una translocación recíproca a nivel génico, con el producto de ARN resultante y la proteína quimérica por la que codificaría. E1, E2, E3, etc. corresponden con los segmentos génicos codificantes (exones). I1, I2, I3, etc. corresponden a los segmentos génicos no codificantes, que separan exones y que suelen ser mucho más extensos que éstos (intrones). El punto de corte de las translocaciones suele encontrarse dentro de segmentos intrónicos.
La caracterización molecular de la presencia de estas translocaciones asociadas a los sarcomas pediátricos puede aportar nuevas herramientas que pueden ser utilizadas en beneficio del paciente. En primer lugar, la presencia de una translocación específica puede confirmar o en algunos casos incluso cambiar la orientación de un diagnóstico basado en criterios histológicos. Por tanto, el diagnóstico molecular puede contribuir al correcto diagnóstico, especialmente en casos ambiguos y/o con un escaso grado de diferenciación. Por otro lado, la presencia o ausencia de translocaciones en algunos casos puede servir como factor pronóstico, motivo por el cual el diagnóstico molecular puede ser un factor adicional a tener en cuenta en la individualización del tratamiento. Además, el producto de una translocación específica puede ser utilizado como marcador altamente específico para la detección de células tumorales circulantes (enfermedad residual mínima) en sangre periférica y médula ósea.
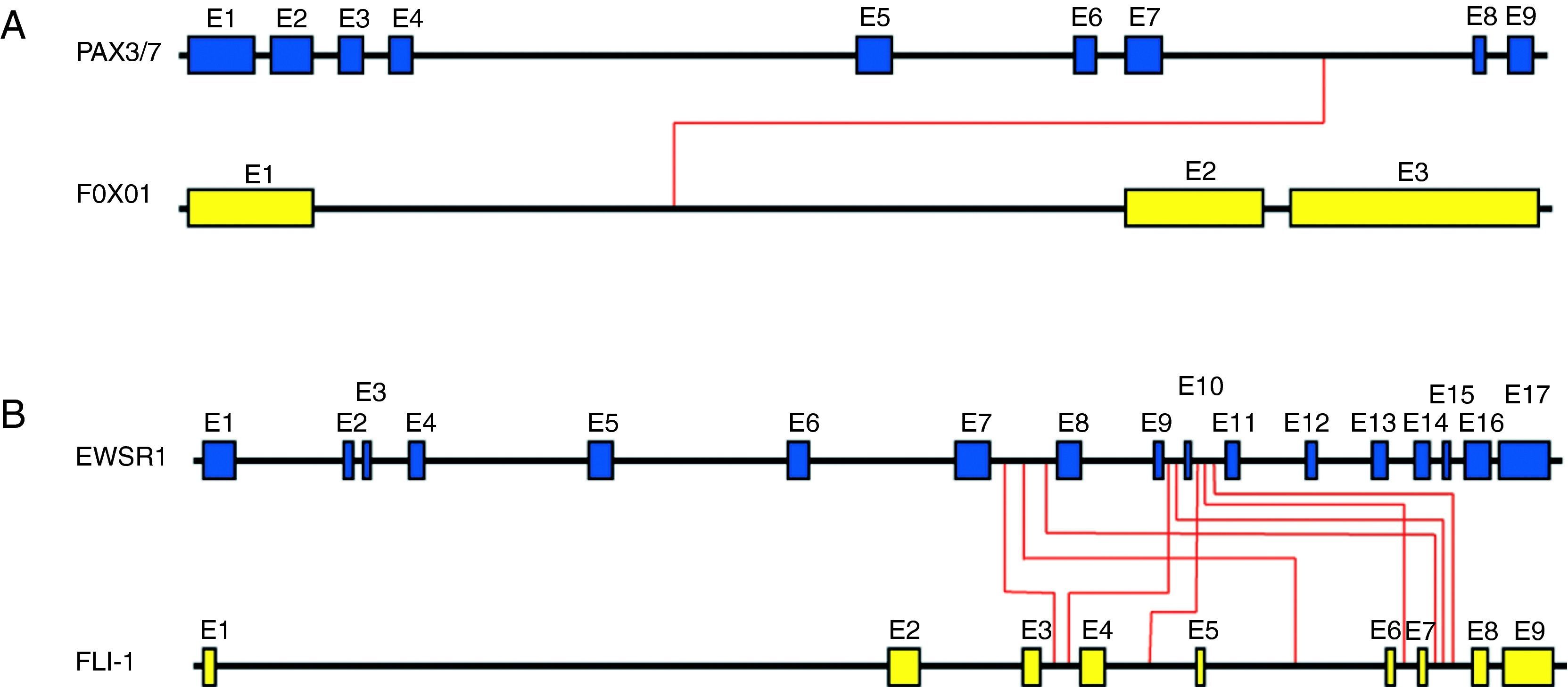
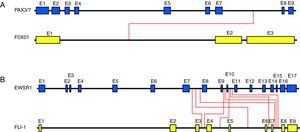
Principales sarcomas pediátricos con alteraciones cromosómicas específicas1-Rabdomiosarcoma (RMS)El rabdomiosarcoma (RMS) es el sarcoma de partes blandas más común en la infancia. Los dos subtipos histológicos principales en los que se subdivide el RMS son el alveolar (ARMS) y el embrionario (ERMS). Para el subtipo embrionario no se han descrito hasta el momento translocaciones específicas. En el subtipo alveolar se han descrito las translocaciones t(2;13) (q35; q14) en aproximadamente un 55% de casos y t(1; 13) (p36; q14) en un 22% de los casos6. Estas translocaciones involucran los genes PAX3 y PAX7, localizados en los cromosomas 2 y 1, respectivamente (fig. 2A). A ambos genes se les ha atribuido un papel importante en el desarrollo embrionario del músculo esquelético y una disrupción de estos genes provoca un desarrollo muscular anormal. En ambos casos la translocación crea un factor de transcripción quimérico con FOXO1 (también denominado FKHR), conservando el dominio de unión a ADN de los genes PAX y adquiriendo el dominio transactivador de FOXO1. La proteína quimérica resultante se une al ADN en los genes diana de PAX correctos, pero su potencia de transactivación es excesiva y no responde a su regulación normal. Se cree que esta desregulación conlleva una alteración biológica muy importante con efectos sobre los mecanismos moleculares que controlan la proliferación, la apoptosis, la diferenciación y la movilidad4,5. Está ampliamente aceptado que el subtipo embrionario tiene mejor pronóstico que el alveolar, por eso la importancia de su correcta identificación. Otra cuestión importante es saber si la presencia de las translocaciones de los genes PAX en el ARMS puede asociarse a un pronóstico desfavorable. Diversos estudios apuntan que la translocación PAX3-FOXO1 está asociada generalmente a un fenotipo más agresivo, pues suele asociarse a tumores de alto grado y, por tanto, a un peor pronóstico. Por el contrario, la translocación PAX7-FOXO1 tiende a darse en individuos más jóvenes, frecuentemente en extremidades, en forma de tumores localizados y asociados a una mejor supervivencia6,7. Además, en los casos de tumores con translocación PAX3-FOXO1 o PAX7-FOXO1, se puede monitorizar la presencia de células tumorales circulantes (enfermedad residual mínima) usando el producto de fusión como marcador altamente específico8,9.
Visión esquemática de ejemplos reales de translocaciones. En la figura se indican los puntos de corte descritos para los genes PAX con FOXO1 (rabdomiosarcoma) (A) y para los genes EWSR1 con FLI-1 (sarcoma de Ewing) (B). Las líneas rojas indican los puntos de corte de las translocaciones conocidas. Los tamaños relativos de los diferentes segmentos génicos son aproximados.
El sarcoma de Ewing (SE) es un tumor maligno del hueso y más raramente de tejido blando que suele presentarse en huesos largos o planos en adolescentes y adultos jóvenes. Es el tumor óseo maligno más frecuente en menores de 20años después del osteosarcoma. Histológicamente, el SE se compone de una población homogénea de células redondas y pequeñas con elevado ratio núcleo/citoplasma. Los sarcomas de la familia Ewing más diferenciados (PNET) pueden presentar cierta diferenciación neuronal. Característicamente, los tumores de esta familia albergan la translocación t(11;22) (q24;q22) que yuxtapone el gen EWSR1 con FLI1 (presente en aproximadamente un 85% de casos) (fig. 2B). También se detecta la translocación EWSR1-ERG (en alrededor del 9-14% de los casos). Un pequeño porcentaje de tumores (entre el 1 y el 5%) presenta también otras translocaciones entre EWSR1 y otros genes relacionados estructuralmente con FLI (EWSR1-ETV1, EWSR1-ETV4 y EWSR1-FEV)10. Diversos hallazgos apuntan que la simple presencia de la translocación bastaría para provocar la transformación oncogénica. En este sentido, se ha demostrado que la expresión de la quimera EWS/FLI es necesaria para el mantenimiento de las propiedades oncogénicas de las células de SE y que su expresión en las células heterólogas NIH3T3 es suficiente para mediar su transformación oncogénica11–13. A pesar de que el rol biológico de la quimera no está del todo esclarecido, se ha demostrado que estos productos de fusión estarían actuando como factores de transcripción anómalos5,14–16 y que podrían estar activando, entre otros, genes activadores del ciclo celular como ciclina D1 y ciclina E, así como regulando a la baja genes inhibidores del ciclo como p21 y p2717,18.
3-Sarcoma sinovial (SS)El sarcoma sinovial (SS) es un sarcoma de partes blandas altamente agresivo, que acaba causando la muerte a más de la mitad de los niños, adolescentes y adultos jóvenes que lo padecen. Es un sarcoma poco común, suponiendo únicamente el 5-10% del total de sarcomas de partes blandas19. Los genes asociados al punto de ruptura son SYT, en el cromosoma 18 y los genes SSX situados en el cromosoma X, SSX1 t(X;18) (p11.2;q11.2), SSX2 t(X;18) (p11.2;q11.2)20,21 y muy raramente SSX4 (X;18) (p11.2;q13)22. Las tres translocaciones son específicas del SS. La translocación se ha descrito en las células fusiformes características de este tumor así como en su componente epitelial23. La presencia de translocaciones entre SYT y los genes SSX en prácticamente todos los casos de sarcoma sinovial sugieren que el producto de la translocación puede tener un papel central en la oncogenicidad de las células que componen este tumor. El gen SYT, también conocido como SS18, parece ser un gen expresado de forma ubicua y la proteína por la que codifica parece tener una función en el remodelado de la cromatina24 así como en la activación transcripcional a través de su interacción con el desacetilador de histonas y represor transcripcional mSin3A25. A pesar de su localización nuclear, las proteínas de la familia SSX no tienen ningún dominio conocido de unión al ADN. No obstante, contienen al menos 2 dominios con actividad de represión transcripcional (dominios KRAP y SSXRD) motivo por el cual se les atribuye una actividad mayoritaria como represores de la transcripción de otros genes, actividad que realizarían a través de la unión a otras proteínas reguladoras de la transcripción como proteínas polycomb, histonas y LHX4 y no a través de la unión directa a ADN26. El conocimiento sobre el comportamiento biológico de la quimera SYT-SSX es todavía bastante limitado, pero el hecho de que esté presente en la práctica totalidad de casos de SS hace pensar que puede estar desarrollando un rol central en la etiología de este tumor. En este sentido, se ha demostrado que la expresión constitutiva del producto quimérico SYT-SSX1 en fibroblastos de rata confiere a estas células propiedades oncogénicas que se demuestran en un aumento de su capacidad proliferativa en cultivo, adquisición de capacidad de proliferación en agar blando y adquisición de la capacidad de formar tumores en ratones inmunodeprimidos27. Además, se ha demostrado que la presencia de SYT/SSX1 promueve la degradación de p53, eliminando por tanto la capacidad supresora de tumores de esta proteína28.
La presencia de la translocación SYT/SSX1 parece estar asociada con la histología bifásica del SS y con un índice de proliferación Ki-67 mayor, mientras que el subtipo monofásico de células fusiformes se asociaría con mayor probabilidad a la presencia de SYT-SSX2 (HaldarM 2008). A pesar de esto, hay cierta controversia sobre una posible correlación entre la presencia de alguna de las isoformas y el pronóstico. Diferentes trabajos apuntan a una mayor mortalidad y/o probabilidad de metástasis para los tumores con la translocación SYT/SSX1 respecto a los que albergan la variante SYT/SSX23,29,30. Por el contrario, otros autores no encontraron ninguna correlación entre el subtipo de SYT/SSX y la supervivencia de los pacientes31–33.
4-Fibrosarcoma congénito infantil (FCI) y nefroma mesoblástico congénito (NMC)A pesar de que la mayor parte de fibrosarcomas se detectan en adultos, entre el 25 y el 40% de los casos se dan en niños menores de 5años, concentrándose especialmente en niños menores de 1año34. El fibrosarcoma congénito infantil presenta en general un fenotipo bastante menos agresivo que el del adulto, aunque la histología es bastante similar en ambos casos35,36. El FCI presenta a nivel histológico similitud con el nefroma mesoblástico congénito (NMC), siendo el curso clínico de ambos sarcomas relativamente benigno, especialmente cuando se comparan con el fenotipo del fibrosarcoma del adulto. En el año 1998 se describió por primera vez en FCI y NMC la misma translocación t(12;15)(p13;q25) que afecta a los genes ETV6 y NTRK3, dando lugar al producto quimérico de fusión ETV6-NTRK337,38. La proteína ETV6, también denominada TEL, es un factor de transcripción de la familia ETS. Activa la transcripción de genes implicados en angiogénesis y hematopoyesis temprana. Se han descrito múltiples translocaciones de ETV6 con otros genes en diversas neoplasias hematopoyéticas. A su vez, la proteína NTRK3 (también denominada TRKC) se localiza en la superficie celular y es un receptor para neurotrofina 3 con actividad kinasa y dependiente de unión a ligando. NTRK3, cuando se une a su ligando, dimeriza y se autofosforila para autoactivarse y fosforilar a su vez otros sustratos. Se conoce que activa, entre otras, vías tan importantes como RAS-RAF39,40, akt/PKB41 y PKC42,43. La quimera ETV6-NTRK3 conserva el dominio de dimerización de ETV6 y el dominio con actividad quinasa de NTRK337. Se cree que este producto quimérico es capaz de dimerizar de forma independiente de ligando y activar constitutivamente la actividad quinasa de NTRK3, conduciendo así a la oncogénesis. En este sentido se ha demostrado que la expresión del producto quimérico en la línea fibroblástica NIH3T3 es suficiente para provocar su transformación oncogénica44. Esta translocación tiene la particularidad de no estar asociada a un solo tipo específico de cáncer, sino que además de detectarse en FSC y NMC, también se ha detectado en carcinoma secretor de mama y en leucemia mielógena aguda45.
5-Tumor miofibroblástico inflamatorio (TMI)El tumor miofibroblástico inflamatorio (TMI) se ha incluido durante largo tiempo bajo la denominación genérica de pseudotumor inflamatorio. El término pseudotumor inflamatorio se ha usado para describir un amplio rango de entidades, que incluyen lesiones neoplásicas y también no-neoplásicas. Durante las dos últimas décadas, el TMI ha emergido como una entidad diferenciada dentro de este grupo, debido a sus particularidades histológicas, clínicas y moleculares. El TMI puede darse a lo largo de toda la vida, pero presenta una clara predilección por su presentación en niños y adolescentes. Puede darse en cualquier parte del cuerpo, aunque las localizaciones más frecuentes son la región abdominopélvica, pulmón y retroperitoneo46.
La presencia de reordenaciones cromosómicas que involucran al gen ALK han sido bien documentadas en TMI, aportando una prueba más a la condición de neoplasia para esta patología. Las reordenaciones de gen ALK fueron descritas originariamente en linfoma anaplásico de células grandes, en el cual aproximadamente el 55% de los pacientes presentaban translocaciones del gen ALK47. En el IMT aproximadamente la mitad de los casos contienen reordenaciones del gen ALK (2p23)48 con diferentes proteínas como TPM3 (1p23), TPM4 (19p13), ATIC (2q35), CLTC (17q23), CARS (11p15), RANBP2 (2q13) y SEC31L1 (4q21)46. La translocación más común detectada en el linfoma anaplásico de células grandes (NPM-ALK) no ha sido detectada nunca en TMI, mientras que TPM3-ALK, ATIC-ALK y CLTC-ALK han sido descritas en ambas neoplasias, representando raros ejemplos de translocaciones compartidas entre neoplasias linfoides y mesenquimales48.
En la última clasificación de la Organización Mundial de la Salud (OMS), se clasifica a los TMI como tumores de potencial biológico intermedio, debido a su tendencia a la recurrencia local pero a un riesgo bastante menor de metástasis. La tasa de recurrencias varía según la localización del tumor, siendo inferior al 2% en tumores pulmonares, llegando aproximadamente hasta un 25% en tumores extrapulmonares. Las metástasis, en cambio, solo se presentan en menos del 5% de los casos46. Diversos estudios apuntan que los pacientes positivos para ALK tendrían un riesgo extremadamente bajo de presentar metástasis por lo que la presencia de alguna translocación podría ser un marcador de buen pronóstico, pero la expresión de ALK no tendría efecto alguno sobre la recurrencia local49,50. En cuanto al diagnóstico diferencial entre TMI y pseudotumor inflamatorio, la presencia de cualquier tipo de translocación en ALK sería muy útil para descartar la segunda opción. Por el contrario, la ausencia de translocaciones en la muestra no permitiría decantarse por ninguna de las dos opciones.
6-Sarcoma alveolar de partes blandas (SAPB)El sarcoma alveolar de partes blandas (SAPB) es un tumor maligno de partes blandas, poco frecuente, con localización predominante en extremidades de adolescentes o jóvenes. A pesar del crecimiento relativamente lento de este tumor, las metástasis pulmonares son frecuentes al diagnóstico.
A nivel molecular, el SAPB se caracteriza por la translocación t(X;17)(p11;p25), descrita en el año 2001 y afectando los genes ASPL y TFE351. ASPL (también denominado ASPSCR1) es un gen ubicuo, de función desconocida, fuertemente expresado en corazón, músculo esquelético, páncreas y testículo, así como en tejidos fetales51. Por el contrario, el gen TFE3 es mucho más conocido. Pertenece a la familia de factores de transcripción hélice-loop-hélice con cremallera de leucinas, dominio proteico que les permite unirse al ADN en forma de homodímeros o heterodímeros para regular la transcripción de determinados genes diana. La quimera ASPL-TFE3 se caracteriza por la pérdida del único dominio conocido de ASPL (dominio UBX) y conserva, por el contrario, los dominios claves de TFE3 para actuar como factor de transcripción, como son el dominio de unión al ADN, el dominio de dimerización y su dominio C-terminal de transactivación52. La naturaleza ubicua de ASPL sugiere una activación constitutiva de su promotor. Por otro lado, el promotor de ASPL es el que queda como regulador de la quimera una vez se ha dado la translocación. Por tanto, la sustitución que se da en el gen translocado de los elementos reguladores de TFE3 por los de ASPL podría dar lugar a una transcripción demasiado activa y desregulada, que podría ser la causa, o al menos una de las causas, de la patogenicidad de la quimera51. Existen dos formas de proteína quimérica según el punto de corte en TFE3, siendo la forma 1 algo más larga que la forma 2. En la forma 2 se pierde el dominio de activación (AD) de TFE3, pero no se ha encontrado ninguna correlación entre el pronóstico y la presencia de una u otra forma de la translocación50,53.
7-Tumor desmoplásico de células redondas y pequeñas (TDCRP)El tumor desmoplásico de células redondas y pequeñas (TDCRP) es una entidad neoplásica muy poco frecuente y muy poco conocida, que fue descrita en 199554. Se caracteriza por una localización predominantemente abdominal del tumor primario, desmoplasia estromal, diferenciación polifenotípica y la presencia de una translocación específica t(11;12)(p13;q12). Aunque puede darse en niños, es más habitual en adolescentes y jóvenes, presentando además cierta preferencia por individuos varones55. El comportamiento biológico del tumor es muy agresivo, con una progresión tumoral rápida y una baja tasa de supervivencia. La translocación, que se presenta de forma recurrente asociada al TDCRP, implica los genes EWSR1 y WT1. Todavía se conoce muy poco de la actividad biológica de la proteína quimérica, pero la expresión de la quimera EWSR1-WT1 vendría regulada por los elementos promotores de EWSR1, mientras que en el extremo 3′ de la quimera, la parte correspondiente a WT1, regularía la expresión génica de algunos factores oncogénicos, entre ellos el PDGF que podría estar relacionado con la fibrosis y la displasia observadas frecuentemente en este tipo de tumor56.
ConclusionesEl diagnóstico de los sarcomas de partes blandas es en muchos casos complejo, debido principalmente a su escasa diferenciación, lo cual dificulta el uso de marcadores proteicos específicos para su correcta clasificación. El conocimiento creciente en el campo de la biología molecular de los sarcomas pediátricos, acumulado principalmente durante las últimas dos décadas, ha abierto la posibilidad de incorporar a la práctica clínica técnicas de diagnóstico molecular basadas en evidencias biológicas. Entre ellas, destacamos en esta revisión por su potencial la detección de reordenaciones cromosómicas, mayoritariamente translocaciones recíprocas. El hecho de tratarse de translocaciones altamente específicas de un tipo concreto de tumor (salvo excepciones) las convierte en herramientas de diagnóstico muy poderosas. Recientemente, incluso se ha propuesto una clasificación de los sarcomas atendiendo a la translocación específica que éstos albergan56. En nuestra unidad, hemos implantado el diagnóstico molecular de translocaciones por PCR desde hace ya unos 5años y los resultados están siendo muy positivos, siendo cada vez más numerosas las muestras externas que recibimos y más los centros que se interesan en el diagnóstico molecular de estas translocaciones.
El conocimiento actual sobre la posible función oncogénica de las translocaciones así como su potencial como factores pronósticos es muy prometedor, aunque en algunos casos es todavía limitado, especialmente en los sarcomas menos frecuentes. En algunos sarcomas, no se han elucidado todavía los mecanismos moleculares de la oncogenicidad de la translocación, ni su posible significación clínica. Por el contrario, el conocimiento es algo más profundo –aunque todavía queda camino por recorrer– en los sarcomas más frecuentes y por tanto mejor conocidos como el rabdomiosarcoma o el sarcoma de Ewing.
El creciente conocimiento sobre la presencia de estas translocaciones asociadas de forma específica a algunos tipos de cáncer, la biología de estas proteínas aberrantes y su más que probable significación clínica está aportando nuevos criterios diagnósticos para los sarcomas de partes blandas. Cada vez más, antes de tomar decisiones clínicas se deberá tener en cuenta el diagnóstico molecular como un parámetro complementario a los criterios clínicos e histopatológicos.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.