La interacción entre factores genéticos y ambientales es clave en el desarrollo de la diabetes tipo 1 (DM1). Aunque las variantes en la región codificante no-HLA son potenciales marcadores para terapias dirigidas, su contribución a la enfermedad es poco conocida. Nuestro objetivo fue caracterizar seis variantes no-HLA en pacientes pediátricos con DM1, analizando su asociación con parámetros clínicos y otras patologías autoinmunes (tiroiditis y enfermedad celiaca [EC]).
Pacientes y métodosSe analizaron las variantes c.1858T>C, c.49A>G, c.919A>G, c.784T>C, c.461G>A y c.-17-6T>A en los genes PTPN22, CTLA4, CD226, SH2B3, FUT2 e INS en una población pediátrica con DM1 (≤18a), comparándola con un grupo control y otro con EC. Las variantes se identificaron mediante PCR cuantitativa usando sondas Taqman.
ResultadosObservamos sobrerrepresentación de las variantes en PTPN22,CD226 e INS en los pacientes con DM1 frente a los grupos con EC y control. Encontramos asociación entre la presencia de autoanticuerpos antiglutamato descarboxilasa (GADA) y la variante en CTLA4 (p=0,005), y entre autoanticuerpos antitirosina fosfatasa asociados a insulinoma (IA2A) y la variante en PTPN22 (p<0,03). El número de autoanticuerpos pancreáticos positivos mostró asociación con la variante en FUT2 (p=0,02). La edad al debut se asoció con las variantes en CTLA4 (p=0,01) y en SH2B3 (p<0,05).
ConclusionesLos pacientes pediátricos con DM1 tienen sobrerrepresentadas las variantes analizadas en los genes PTPN22, CD226 e INS, señalando posibles dianas terapéuticas que modulen el proceso autoinmune. Su asociación con determinados perfiles clínicos y autoinmunes podría ayudar a identificar pacientes de riesgo y optimizar su seguimiento.
The interplay between genetic and environmental factors plays a critical role in the development of type 1 diabetes (T1D). While variants in non-HLA coding regions have been identified as potential therapeutic targets, their exact contribution to the pathophysiology of disease remains unclear. The aim of the study was to characterize six non-HLA variants in pediatric patients with T1D and explore their potential association with clinical parameters and other autoimmune diseases, such as thyroiditis and celiac disease (CD).
MethodsWe analyzed six variants (c.1858T>C, c.49A>G, c.919A>G, c.784T>C, c.461G>A, and c.-17-6T, located in the PTPN22, CTLA4, CD226, SH2B3, FUT2 and INS genes) in a pediatric sample (age ≤ 18 years) with T1D, comparing it with a CD group and a control group. The variants were genotyped using quantitative PCR with TaqMan probes.
ResultsWe observed that variants in PTPN22, CD226 and INS were overrepresented in patients with T1D compared to the control and CD groups. There was a significant association between the presence of anti-glutamate decarboxylase autoantibodies (GADA) and the CTLA4 variant (P=.005), as well as between insulinoma-associated anti-tyrosine phosphatase autoantibodies (IA2A) and the PTPN22 variant (P <.03). The number of positive pancreatic autoantibodies was associated with the FUT2 variant (P=.02). Additionally, age at onset was associated with CTLA4 (P=.01) and SH2B3 (P <.05) variants.
ConclusionThe analyzed variants in the PTPN22, CD226, and INS genes were overrepresented in pediatric patients with T1D, suggesting potential therapeutic targets for modulating the autoimmune process. Their associations with specific clinical and autoimmune profiles can be applied in the identification of high-risk patients and help optimize their follow-up.
La diabetes tipo 1 (DM1) es la patología metabólica crónica más frecuente en la edad pediátrica y se caracteriza por la presencia de hiperglucemia secundaria a un déficit de insulina, debido a la destrucción inmunomediada de las células β del páncreas1. Según datos de la International Diabetes Federation, en 2023, España fue uno de los diez países con mayor prevalencia de DM1 en todas las edades, contabilizando 17.245 pacientes con DM1 menores de 20 años2. Datos actuales apoyan un incremento anual de su incidencia de un 2,5%3, siendo una de las enfermedades más prevalentes y con mayor impacto en la población infantil. Se predice, además, un aumento significativo de los casos para 2040, especialmente en países en desarrollo4.
Los principales biomarcadores de la DM1 son los autoanticuerpos (Acs) antiglutamato descarboxilasa (GADA), Acs antitirosina fosfatasa asociados a insulinoma (IA2A) y Acs antiinsulina (IAA)5. Recientemente, se han descrito los anticuerpos anti transportador 8 del zinc (anti-ZnT8), que se han generalizado como marcador adicional en la caracterización de la diabetes tipo 16. Además, la DM1 presenta una frecuente asociación con otras enfermedades autoinmunes, como la tiroiditis y la enfermedad celiaca (EC)7.
La DM1 es una enfermedad poligénica, en la que la interacción entre factores genéticos y ambientales desempeña un papel crucial en el desarrollo del proceso autoinmune. Aproximadamente la mitad del riesgo genético total se atribuye a variantes HLA de clase II y del gen de la insulina (INS), asociadas con una desregulación de la autoinmunidad mediada por linfocitos T7, mientras que la otra mitad se ha atribuido a variantes no-HLA8. Esta distribución refuerza la pertinencia de estudiar las variantes no-HLA para comprender la predisposición genética a la DM1.
En los últimos años se han llevado a cabo distintos estudios de asociación del genoma completo (Genome-Wide Association Studies [GWAS]), que han permitido la identificación de múltiples variantes genéticas de riesgo no-HLA, que parecen estar relacionadas con la heterogeneidad clínica de la enfermedad9,10. Se han identificado más de 90 alelos de riesgo asociados con esta enfermedad11. A pesar de que muchas de estas variantes de riesgo se localizan en regiones intrónicas y, por lo tanto, no son codificantes9, un pequeño porcentaje de ellas se localizan en la región codificante, pudiendo llegar a ser potenciales marcadores para terapias dirigidas. Una reciente revisión de Shapiro et al.10 analizó 13 polimorfismos de nucleótido único (Single Nucleotide Polimorphisms [SNPs]) codificantes en 10 genes diferentes (PTPN22, IFIH1, SH2B3, CD226, TYK2, FUT2, SIRPG, CTLA4, CTSH y UBASH3A), describiendo no solo la relación de estas variantes con el riesgo de desarrollar DM1, sino también sus potenciales implicaciones clínicas y terapéuticas dentro de la medicina de precisión. Estas variantes modulan distintos momentos de la autoinmunidad pediátrica: por ejemplo, la variante en CTLA4 se asocia con la rápida progresión de la autoinmunidad11, mientras que la variante en SH2B3 podría estar vinculada con el desarrollo inicial de Acs12. Además, muchas de estas variantes identificadas en DM1 se han relacionado con otras patologías autoinmunes, como tiroiditis y EC, lo que resalta la agregación de autoinmunidades en la población pediátrica13. Esta coexistencia tiene un valor clínico y traslacional, ya que las terapias dirigidas frente a estas variantes podrían ser de utilidad en múltiples enfermedades.
El desarrollo reciente de nuevas terapias capaces de frenar, al menos temporalmente, la progresión a fases clínicas de la enfermedad, ha puesto de manifiesto la necesidad de realizar un diagnóstico precoz y preciso. Un enfoque para convertir los datos genéticos en una medida predictiva de la susceptibilidad a la enfermedad consiste en sumar los efectos de riesgo, tanto de las variantes HLA como de polimorfismos no HLA, en un score de riesgo poligénico.
Con estos antecedentes, decidimos caracterizar cinco alelos de riesgo (rs2476601, rs231775, rs763361, rs3184504 y rs601338) localizados en regiones codificantes de los genes no-HLA PTPN22, CTLA4, CD226, SH2B3 y FUT2, y una variante intrónica en el gen de la insulina (INS), en una cohorte de pacientes pediátricos diagnosticados con DM1. Los objetivos de este estudio son estimar la prevalencia de las variantes genéticas analizadas en la población a estudio y compararla con una población control, así como analizar su posible asociación con parámetros clínicos (edad de debut, autoanticuerpos de DM1 al debut) y otras enfermedades autoinmunes (tiroiditis y EC). Esto permitirá evaluar la posible relación de estas variantes con la variabilidad clínica observada en estos pacientes, así como identificar potenciales marcadores para terapias dirigidas que puedan mejorar la calidad de vida de estos pacientes dentro de un enfoque de medicina personalizada.
Pacientes y métodosSe trata de un estudio observacional, retrospectivo, analítico y transversal, basado en el análisis de variantes genéticas no-HLA de riesgo para DM1 en una cohorte de pacientes pediátricos (≤ 18 años) diagnosticados de esta patología, y en seguimiento en un hospital de tercer nivel. Dado que las variantes genéticas analizadas están descritas en otras patologías autoinmunes, como la EC, analizamos también la prevalencia de dichas variantes en una serie de pacientes con EC sin DM1, procedentes de gastroenterología pediátrica del mismo centro. Además, incluimos una población control de pacientes pediátricos de nuestro hospital, sin patologías autoinmunes, cuyas frecuencias alélicas eran similares a las reportadas para la población caucásica en la base de datos Reference SNP Report (National Institute of Health).
Los pacientes con DM1 incluidos en el estudio fueron reclutados en la consulta de Endocrinología y Diabetes Pediátrica de un hospital de tercer nivel y firmaron un consentimiento informado de acuerdo con la normativa vigente, el cual fue aprobado por su Comité de Ética. Se excluyeron aquellos pacientes sin consentimiento informado, aquellos de los que no se obtuvo muestra suficiente para el estudio o resultó imposible realizar la venopunción, así como pacientes con cromosomopatías (síndrome de Down, síndrome de Turner…) y poliendocrinopatías familiares. Para el análisis de los pacientes con EC y del grupo control se utilizó el excedente de muestra de los análisis de seguimiento rutinarios.
Los datos clínicos de los pacientes se recogieron en una base de datos creada para el estudio utilizando la historia clínica electrónica. Se incluyeron la edad al debut de la DM1, el número de autoanticuerpos positivos (GADA, IA2A e IAA), la presencia o ausencia de anticuerpos antitiroideos (anti-TPO –anticuerpos antiperoxidasa– y/o anti-TG –anticuerpos antitiroglobulina–) y la presencia o ausencia de EC y/o hipotiroidismo.
Los Acs pancreáticos se analizaron mediante la técnica de ensayo inmunoadsorbente ligado a enzima (ELISA) en el Triturus (Grifols), mientras que los anticuerpos antitiroideos se analizaron mediante inmunoensayo quimioluminiscente en el Atellica® Solution (Siemens, Healthineers, Múnich, Alemania).
Para la obtención de las muestras se utilizó una muestra de sangre total anticoagulada en tubo con K3-EDTA. El ADN fue extraído utilizando un sistema QIAamp® DNA Mini Kit (Qiagen, Hilden, Alemania) para su posterior cuantificación en un espectrofotómetro NanoDropTM (Thermo Fisher Scientific, Waltham, Massachusetts, Estados Unidos). Por último, las muestras de ADN fueron almacenadas en criotubos de 2mL a -80°C hasta su utilización.
La identificación de las variantes se llevó a cabo utilizando 5 sondas Taqman prediseñadas para las variantes en los genes PTPN22 (C__16021387_20), CTLA4 (C___2415786_20), CD226 (C___1464836_20), SH2B3 (C___2981072_10), INS (C___1223317_10) y una sonda de diseño customizado para la variante en el gen FUT2 (ANWDEJA) de Thermofisher Scientific en un sistema de PCR cuantitativa Step One Plus (Applied Biosystems, Foster City, California, Estados Unidos) (tabla 1).
Variantes no HLA de riesgo a analizar en el estudio
| Ensayo | Gen | Identificación rs | Cambio |
|---|---|---|---|
| C__16021387_20 | PTPN22 | rs2476601 | p.Trp620Argc.1858T>C |
| C___2415786_20 | CTLA4 | rs231775 | p.Thr17Alac.49A>G |
| C___1464836_20 | CD226 | rs763361 | p.Ser307Glyc.919A>G |
| C___2981072_10 | SH2B3 | rs3184504 | p.Trp262Argc.784T>C |
| ANWDEJA | FUT2 | rs601338 | p.Trp154Terc.461G>A |
| C___1223317_10 | INS | rs689 | c.-17-6T>A |
PTPN22: gen de la proteína tirosina fosfatasa no receptora de tipo 22; CTLA4: gen del antígeno 4 asociado a los linfocitos T citotóxicos; CD226: gen de la molécula del clúster de diferenciación 226; SH2B3: gen de la proteína 3 adaptadora SH2B; FUT2: gen que codifica la α(1,2)-fucosiltransferasa 2; INS: gen de la insulina.
La frecuencia alélica se comparó tanto con una población control de nuestro centro (n=50 con edad ≤ 18 años) como con la frecuencia alélica poblacional de la base de datos Reference SNP Report (National Institute of Health) para raza caucásica, donde estaba incluida el 86,7% de nuestra probación de estudio.
Para el estudio descriptivo y estadístico, las variables cuantitativas se expresaron como mediana (rango intercuartílico). Los test estadísticos utilizados fueron el test de chi-cuadrado, con el fin de determinar si existía elación entre cada una de las variantes no-HLA y las variables clínicas; la prueba de Kruskal-Wallis, test no paramétrico mediante el que se establece la distribución de la edad al debut de la enfermedad en relación con cada variante, y la prueba U de Mann-Whitney cuando se trataba de estudiar la relación entre dos grupos independientes. Para comparar las frecuencias alélicas se utilizó la prueba de chi-cuadrado, y para frecuencias alélicas muy bajas se utilizó el test exacto de Fisher. El análisis estadístico se realizó mediante el Stata v15.1 Software (StataCorp2017, StataCorp, College Station, Texas, Estados Unidos). Las pruebas con un valor de p<0,05 se consideraron estadísticamente significativas.
ResultadosDel total de 194 pacientes pediátricos (≤18 años) con DM1, 94 eran mujeres y 99 varones. La mediana de la edad de debut fue 8,3 años (IQR: 4,5-10,9), sin diferencias significativas por sexo. De los 189 pacientes con datos de autoinmunidad pancreática (GADA, IA2A e IAA), 168 tuvieron algún anticuerpo positivo (88,83%) y 21 resultaron negativos para los tres Acs analizados. De los 186 pacientes con información de autoinmunidad tiroidea, 37 presentaron autoanticuerpos positivos (aTPO o aTG) y nueve de ellos desarrollaron hipotiroidismo. Además, 16 de los 194 fueron diagnosticados de EC (8,2%) y cinco de ellos asociaron tanto anticuerpos tiroideos como EC. Cabe aclarar que la positividad de los Acs se consideró al momento del diagnóstico de DM1 y que las enfermedades autoinmunes reportadas fueron clínicamente confirmadas.
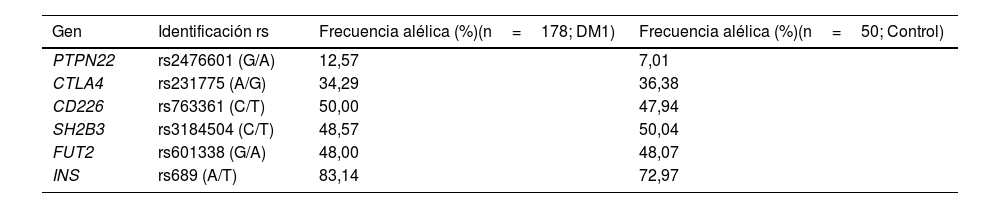
Las frecuencias alélicas, tanto en el grupo de pacientes con DM1 como en el grupo control, se muestran en la tabla 2. Se observó una sobrerrepresentación de las variantes en PTPN22, CD226 e INS en pacientes con DM1, objetivándose significación estadística en las variantes en CD226 (p=0,041) e INS (p=0,019).
Frecuencia de los alelos de riesgo en pacientes con DM1 y en el grupo control
| Gen | Identificación rs | Frecuencia alélica (%)(n=178; DM1) | Frecuencia alélica (%)(n=50; Control) |
|---|---|---|---|
| PTPN22 | rs2476601 (G/A) | 12,57 | 7,01 |
| CTLA4 | rs231775 (A/G) | 34,29 | 36,38 |
| CD226 | rs763361 (C/T) | 50,00 | 47,94 |
| SH2B3 | rs3184504 (C/T) | 48,57 | 50,04 |
| FUT2 | rs601338 (G/A) | 48,00 | 48,07 |
| INS | rs689 (A/T) | 83,14 | 72,97 |
DM1: diabetes tipo 1; PTPN22: gen de la proteína tirosina fosfatasa no receptora de tipo 22; CTLA4: gen del antígeno 4 asociado a los linfocitos T citotóxicos; CD226: gen de la molécula del clúster de diferenciación 226; SH2B3: gen de la proteína 3 adaptadora SH2B; FUT2: gen que codifica la α(1,2)-fucosiltransferasa 2; INS: gen de la insulina.
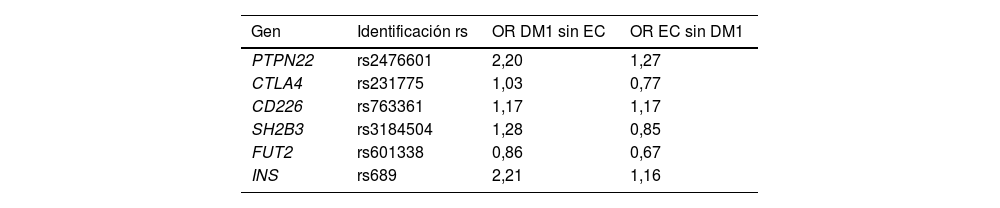
Las odds ratios (OR) de las distintas variantes genéticas quedan resumidas en la tabla 3, reflejando una sobrerrepresentación de algunas de ellas en el grupo de pacientes con DM1 sin EC, comparado con el grupo de EC. Estos OR no están ajustados por edad, sexo ni genotipos HLA.
Variantes de riesgo a analizar en el estudio con sus respectivos odds ratio (OR) o razón de probabilidades en pacientes con diabetes tipo 1 sin enfermedad celiaca (DM1 sin EC) y con enfermedad celiaca sin diabetes tipo 1 (EC sin DM1)
| Gen | Identificación rs | OR DM1 sin EC | OR EC sin DM1 |
|---|---|---|---|
| PTPN22 | rs2476601 | 2,20 | 1,27 |
| CTLA4 | rs231775 | 1,03 | 0,77 |
| CD226 | rs763361 | 1,17 | 1,17 |
| SH2B3 | rs3184504 | 1,28 | 0,85 |
| FUT2 | rs601338 | 0,86 | 0,67 |
| INS | rs689 | 2,21 | 1,16 |
PTPN22: gen de la proteína tirosina fosfatasa no receptora de tipo 22; CTLA4: gen del antígeno 4 asociado a los linfocitos T citotóxicos; CD226: gen de la molécula del clúster de diferenciación 226; SH2B3: gen de la proteína 3 adaptadora SH2B; FUT2: gen que codifica la α(1,2)-fucosiltransferasa 2; INS: gen de la insulina.
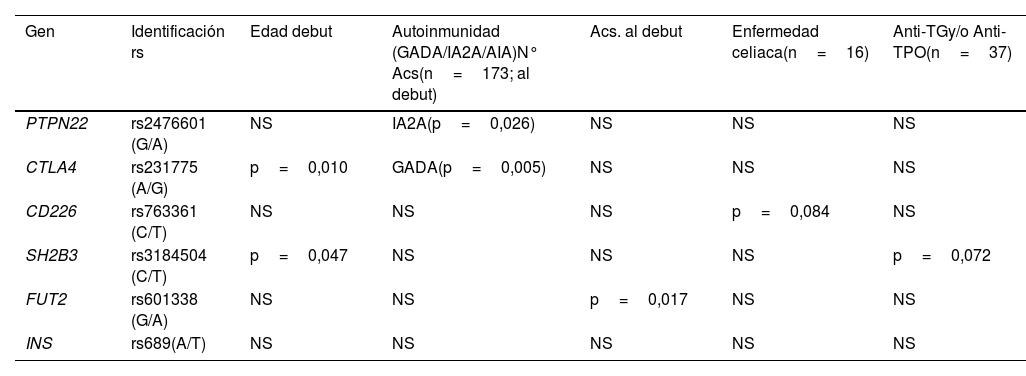
La asociación entre las distintas variantes genéticas analizadas con el perfil clínico queda resumida en la tabla 4. Se encuentra asociación entre la presencia de GADA y la variante en CTLA4 (p=0,01), así como entre IA2A y la variante en PTPN22 (p<0,03). La edad al debut se asoció con las variantes de CTLA4 (p=0,010) y SH2B3 (p<0,05). También se observó asociación entre la variante de FUT2 (p=0,017) y un mayor número de Acs pancreáticos al debut. No se detectó asociación entre la variante en CD226 y la EC (p=0,084) ni entre la variante en SH2B3 y la presencia de anticuerpos antitiroideos (p=0,072).
Asociación entre las distintas variantes genéticas y el perfil clínico y autoinmune en el grupo de pacientes con diabetes tipo 1 sin enfermedad celiaca
| Gen | Identificación rs | Edad debut | Autoinmunidad (GADA/IA2A/AIA)N° Acs(n=173; al debut) | Acs. al debut | Enfermedad celiaca(n=16) | Anti-TGy/o Anti-TPO(n=37) |
|---|---|---|---|---|---|---|
| PTPN22 | rs2476601 (G/A) | NS | IA2A(p=0,026) | NS | NS | NS |
| CTLA4 | rs231775 (A/G) | p=0,010 | GADA(p=0,005) | NS | NS | NS |
| CD226 | rs763361 (C/T) | NS | NS | NS | p=0,084 | NS |
| SH2B3 | rs3184504 (C/T) | p=0,047 | NS | NS | NS | p=0,072 |
| FUT2 | rs601338 (G/A) | NS | NS | p=0,017 | NS | NS |
| INS | rs689(A/T) | NS | NS | NS | NS | NS |
PTPN22: gen de la proteína tirosina fosfatasa no receptora de tipo 22; CTLA4: gen del antígeno 4 asociado a los linfocitos T citotóxicos; CD226: gen de la molécula del clúster de diferenciación 226; SH2B3: gen de la proteína 3 adaptadora SH2B; FUT2: gen que codifica la α(1,2)-fucosiltransferasa 2; INS: gen de la insulina; Acs: autoanticuerpos; Anti-TG: anticuerpos antitiroglobulina; Anti-TPO: anticuerpos antiperoxidasa; GADA: autoanticuerpos antiglutamato descarboxilasa; IAA: autoanticuerpos antiinsulina;, IA2A: autoanticuerpos antitirosina fosfatasa asociados a insulinoma; NS: no significativo.
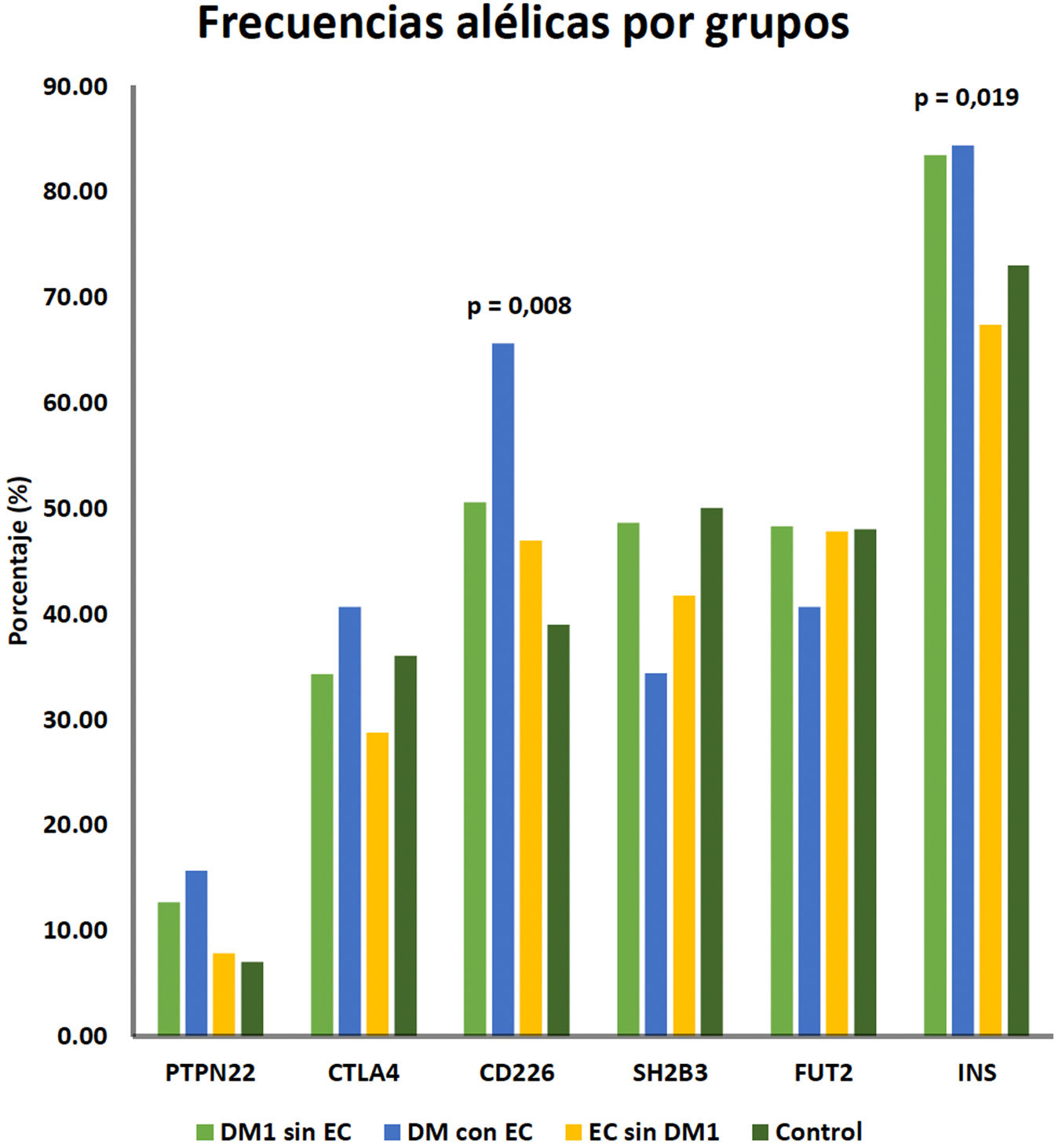
Además de los 194 pacientes pediátricos reclutados con DM1 (16 de ellos con DM1+EC), se analizaron 115 pacientes con EC sin DM1 (63 mujeres y 52 varones). La mediana de la edad de diagnóstico fue 11,3 años (IQR: 7,57-14,4). El total de los pacientes se agrupó en cuatro categorías: DM1 sin EC, DM1 con EC, EC sin DM1 y grupo control. La comparación de las frecuencias alélicas de las variantes analizadas en estos grupos se muestra en la figura 1. Se observó una sobrerrepresentación de las variantes en PTPN22, CD226 e INS en pacientes con DM1, comparados con los otros grupos. El análisis estadístico reveló diferencias estadísticamente significativas en las variantes en CD226 (p=0,041) e INS (p=0,019) al comparar el grupo DM1 sin EC con el grupo control. Además, al comparar el grupo de pacientes con DM1 y EC con el grupo control se observó significación estadística en la variantes en CD226 (p=0,008). Por el contrario, no se encontraron diferencias estadísticamente significativas entre el grupo de pacientes con EC sin DM1 y el grupo control, ni entre los grupos DM sin EC y DM con EC.
Frecuencias alélicas de las variantes genéticas analizadas en los distintos grupos. DM1 sin EC: grupo con diabetes tipo 1 sin enfermedad celiaca; DM1 con EC: grupo con diabetes tipo 1 y enfermedad celiaca; EC sin DM1: grupo con enfermedad celiaca sin diabetes tipo 1; control: población control.
Los resultados obtenidos concuerdan con los hallazgos de otros estudios, incluyendo estudios de gran magnitud como TEDDY y TrialNet10,14-18, que describen variantes no-HLA de riesgo para el desarrollo de DM1, habiendo observado una sobrerrepresentación de algunas de ellas (genes INS, PTPN22 y CD226) en nuestro grupo de pacientes pediátricos con DM1. De forma significativa, objetivamos una frecuencia alélica diferencial de algunas de estas variantes en los distintos grupos (DM1 sin EC, DM1 con EC, EC sin DM1 y grupo control) (fig. 1). Destacar que la frecuencia detectada de EC y DM1 (8,2%) es similar a la descrita por otros autores19. Estos hallazgos respaldan el carácter poligénico de la enfermedad, además de señalar posibles dianas terapéuticas que modulen el proceso autoinmune. Por otro lado, la sobrerrepresentación de algunas variantes, como la observada en el gen CD226 en pacientes con más de una patología autoinmune (DM1 y EC), podría señalar un perfil de susceptibilidad distintivo en un subgrupo de pacientes.
De este modo, la variante más común en el gen PTPN22 es la rs2476601, que presenta una adenina (A) en lugar de la base nitrogenada mayoritaria guanina (G), y da lugar al cambio p.Arg620Trp. Esta variante se considera el factor de riesgo más significativo para el desarrollo de DM1 después del HLA-DR/DQ y del gen INS, con un OR de 1,8110. En nuestro estudio hemos encontrado dicha variante sobrerrepresentada en los pacientes con DM1 comparado tanto con la población control analizada, como frente al grupo de EC sin DM1. Según los resultados de los estudios GWAS, la presencia de esta variante parece estar relacionada con la aparición de IAA en estadios iniciales15, así como con otras patologías autoinmunes y la aparición temprana de la enfermedad16,17. Aunque en nuestra población no hemos observado dicha asociación, sí que objetivamos una relación entre esta variante y la presencia de IA2A, descritos en estadios en los que ya hay destrucción de la célula β pancreática, lo cual no se había identificado previamente. Ello puede deberse a un distinto estadio evolutivo de nuestra cohorte frente a las descritas, o bien a diferencias poblacionales.
De forma similar, la variante rs231775 en el gen CTLA4, en la que el cambio A por G genera una sustitución p.Thr17Ala, parece estar relacionada con la presencia de GADA y el inicio tardío de la enfermedad20,21. En nuestro estudio confirmamos la asociación entre la presencia de GADA y una mayor edad al debut con esta variante. La implicación de este receptor CTLA-4 en la regulación de las células T ha favorecido el interés en el uso del fármaco abatacept, proteína de fusión CTLA4-IgG1 que bloquea la activación de los linfocitos T, para retrasar la pérdida de función de las células β en pacientes recién diagnosticados de DM122. Así, existen en la actualidad varios ensayos clínicos que pretenden evaluar la utilidad de esta molécula, ya sea sola o en combinación con rituximab (anti-CD20) para prevenir o retrasar la progresión de la DM1 en estadios iniciales23.
La variante rs3184504 en el gen SH2B3 presenta un cambio de C por T que genera una sustitución R262W. Esta variante se ha asociado en otros estudios principalmente con el desarrollo de Acs frente a la célula beta pancreática12. En nuestra serie, hemos encontrado asociación entre dicha variante y una mayor edad de inicio de la enfermedad. Además, encontramos que esta variante estaba menos expresada en pacientes con EC, asociada o no con DM1, esto podría estar relacionado con su caracterización como posible factor protector para EC, reduciendo el riesgo de desarrollar esta enfermedad11.
Por último, en relación con la variante rs601338 en FUT2 que implica una sustitución de G por A, dando lugar al cambio p.W154*, observamos una asociación significativa con la presencia de al menos un autoanticuerpo al debut. La detección de esta variante y su relación con los Acs al debut es importante, ya que en estudios previos se ha observado un mayor deterioro en la primera fase de respuesta de la insulina al test de sobrecarga intravenosa de glucosa, en niños con múltiples Acs y con esta variante específica24. Además, su relevancia clínica radica, según algunos autores, en que su presencia en pacientes con DM1 aumenta la susceptibilidad a alteraciones de la microbiota intestinal y el estado inflamatorio, afecciones que pueden mitigarse con intervenciones tempranas como dietas adaptadas, prebióticos, probióticos o leche de fórmula enriquecida con α1,2-fucosil-oligosacáridos25.
Como principal limitación del estudio, destaca el limitado número de pacientes en los diferentes subgrupos, como aquellos con DM1 y EC o con DM1 e hipotiroidismo, debido a la prevalencia de dichas patologías en población pediátrica. Asimismo, el análisis genético incluyó un conjunto limitado de variantes, sin abarcar todas las descritas previamente por otros autores. Otra limitación es que no se estudió la relación de las variantes con los anti-ZnT8. Otra posible limitación es que algunas variantes con baja prevalencia no se identifiquen en un número suficiente de pacientes para lograr resultados estadísticamente significativos. Para abordar este desafío, nuestro grupo continúa aumentando el tamaño de la muestra. Además, subrayamos la necesidad de llevar a cabo estudios multicéntricos con una mayor potencia para confirmar de manera concluyente nuestros resultados.
Cabe resaltar que la identificación de los genes que presentan variantes de riesgo puede ayudarnos a orientar la terapia más adecuada al perfil de cada paciente, incluyendo también fórmulas de estimación de riesgo26-28. De esta forma, aquellos pacientes portadores de variantes de riesgo en los genes PTPN22, CD226 y CTLA4 podrían beneficiarse de fármacos moduladores de la coestimulación de células T10. De forma similar, existe un interés en las terapias de tolerancia antígeno-específica en pacientes con variantes de riesgo en PTPN22 y CTLA4, por su propensión a desarrollar anticuerpos IAA y GADA, respectivamente, en estadios iniciales15.
Aunque las variantes no-HLA identificadas son prometedoras como biomarcadores, su utilidad clínica individual sigue siendo limitada y requiere validación en cohortes independientes y multicéntricas.
ConclusionesLa sobrerrepresentación detectada en los pacientes pediátricos con DM1, de algunas de las variantes analizadas en los genes PTPN22, CD226 e INS para el desarrollo de dicha enfermedad, apoyan el carácter poligénico de la misma y señalan posibles dianas terapéuticas que modulen el proceso autoinmune.
La asociación detectada entre algunas variantes de riesgo no-HLA y determinados perfiles clínicos y autoinmunes, así como con la edad al debut, podría ayudar a identificar pacientes de riesgo y guiar el seguimiento clínico.
La sobrerrepresentación de algunas variantes (en CD226) en pacientes con más de una patología autoinmune (DM1 y EC) podría señalar un perfil de susceptibilidad distintivo en un subgrupo de pacientes.
Futuros estudios deberían incluir un mayor tamaño muestral y el análisis de otras variantes genéticas no-HLA asociadas a la DM1 y otras patologías autoinmunes. También sería de interés profundizar en el perfil alélico y autoinmune de los hermanos de pacientes con DM1, con o sin EC, así como analizar los HLA de clase II de riesgo en DM1, para calcular scores de riesgo que combinen variantes HLA, no-HLA y otros perfiles clínicos y de riesgo previamente identificados. Adicionalmente, la comparación de las variantes identificadas en el grupo minoritario de pacientes en los que no se detectan Acs con las del grupo de pacientes que sí los presentan, podría aportar información sobre diferencias que puedan explicar el desarrollo de la patología.
FinanciaciónEste trabajo ha sido parcialmente financiado mediante la Beca José Igea 2023.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.